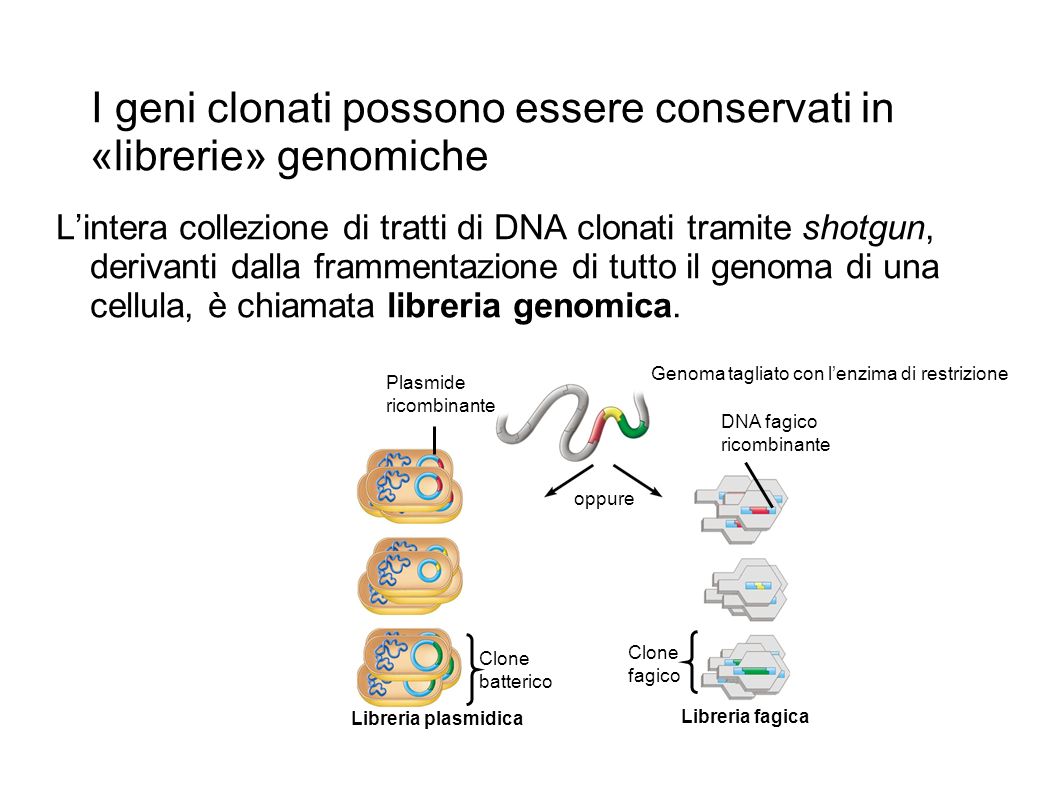
PLFA
Il polimorfismo di lunghezza del frammento Amplificato (PLFA) è una variante della tecnica di fingerprinting genetico (DNA fingerprinting) basato sull’amplificazione selettiva mediante reazione a catena della polimerasi (PCR) frammento di digestione del DNA genomico con una coppia di enzimi restrizione. Vedere impronte digitali del DNA. Il metodo originale di Pieter Vos et al. Si compone di tre fasi:
1) digestione del DNA con una coppia di enzimi di restrizione (es EcoRI e MseI) e legatura oligonucleotidi a doppio filamento (adattatori). Verso le estremità dei frammenti prodotti.
2) amplificazione selettiva di un insieme di quei frammenti da una coppia di primers complementari verso gli adattatori e le sequenze di riconoscimento di enzimi di restrizione (un primer alle estremità scisso con EcoRI e il secondo primer per le estremità scisso con MseI). Gli inneschi hanno tre nucleotidi rimanenti a sua estremità 3′(il nucleotide ‘selettivo’) che riconoscono solo (e consente di amplificare) frammenti di restrizione avente i corrispondenti tre nucleotidi complementari che fiancheggiano il sito di restrizione, riducendo (1/16 per ogni nucleotide selettivo) il numero di frammenti amplificati;
3) analisi dei frammenti amplificati in un gel di elettroforesi. Questo metodo può generare un’impronta genetica da qualsiasi campione di DNA, indipendentemente, dall’origine o complessità, senza una preventiva conoscenza di qualsiasi sequenza senza gli svantaggi di altri metodi di tipizzazione che sono estremamente sensibili alle condizioni di reazione, qualità del DNA e delle variazioni di temperatura. Permette anche l’amplificazione simultanea di un gran numero di frammenti di restrizione (generalmente da 50 a 100) in ogni analisi eseguita.