Materiale richiesto per PCR
Pcr materiale ed equipaggiamento e materiali richiesti:
- Dna stampo (0.1 g/ μl ),
- Primer diretto (10 M ),
- Primer opposto (10 M ),
- Taq Dna polimerasi ( 5U/ μl ),
- Tampone 10x,
- Mix di dNTP (10 mM di ciascun dNTP ),
- MgCl2 25 mM, cofattore per la Taq,
- Acqua priva di nucleasi,
- Termociclatore,
- Pipette regolabili e microprovette eppendorf,
- Centrifuga per microprovette.
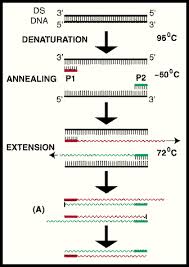
Amplificazione per PCR
Amplificazione, Note:
- usare puntali individuali per tutte le addizioni al fine di evitare la cross-contaminazione del campione.
- combinare i primi sei componenti in ordine di lista nella tabella 1 in una microprovetta di reazione di 0.2 ml. Brevemente centrifugare in una microcentrifuga.
- Iniziare la reazione addizionando il Dna stampo e la Taq Dna polimerasi.
- Mettere le provette in un sistema a temperatura controllata, blocca calore, e procedere nel termociclatore col profilo scelto per le vostre reazioni.
Thermal cycling profile per PCR
Temperatura pre-denaturazione 96°C 5min,
Denaturation temperature 94°C 30 sec,
Anneal temperature 45-65°C 30 sec,
Extension temperature 72°C 60 sec,
Post extension temperature 72°C 7 min,
4°C hold
Analisi
• Dopo che la PCR è stata completata (la maggior parte dei programmi di PCR si aggira intorno alle due ore ), analizzare i prodotti di reazione mediante elettroforesi su gel d’agarosio prelevando un’aliquota di 5 μl dalla reazione totale. I prodotti saranno resi visualizzabili dalla trans-illuminazione UV del gel colorato col bromuro d’etidio.
• I prodotti di reazione possono essere conservati a -20°C.
• Un minuto è necessario per estendere 1Kb.
PCR polimerase chain reaction
La PCR, polimerase chain reaction (reazione a catena della polimerasi) è una tecnica di clonazione,ma per realizzarla bisogna conoscere le estremità della sequenza che vogliamo amplificare.
Requisiti dei primers:
- Lunghezza ≈ 20 basi ( = > di 17 nucleotidi ).
- Contenuto G/C ≈ 45-55%.
- Temperatura di melting.
- Temperatura di annealing.
Problemi che possono insorgere relativamente ai primers
- Un primer può essere self complementare e ripiegarsi in un hairpins.
- L’estremità 3’ del primer è appaiato impedendo l’annealing.
- Un primer può formare un dimero con se stesso o un altro primer.
Stima delle temperature di melting e di annealing dei primers.
Temperatura di melting dei primers
Calcolo delle temperature di melting dei primers:
• Tm = 4° gradi di temperatura per la somma delle C e delle G più 2° gradi di temperatura per la somma delle A e delle T
4°x( C+G ) + 2°x( A+T )
Temperatura di annealing dei primers
Calcolo della temperatura di annealing dei primers:
Ta = Tm2
Verifica del risultato di una PCR, L’etidio bromuro dà una colorazione rosa fluorescente visualizzabile sotto U.V.
- Misura del frammento.
- Presenza di più di una banda (prodotto aspecifico)
Se si verifica ciò bisogna ottimizzare la PCR regolando la temperatura di annealing. Al riguardo si fanno diverse prove di PCR a diverse temperature o andando a regolare la concentrazione di ioni Mg++( 1 – 4 mM ).
Il Mg++ è indispensabile per l’attività della Taq polimerasi e la sua concentrazione è molto importante. Infatti se la concentrazione del magnesio che sta tra 1 e 4 mM è inferiore a questi parametri si ha una resa bassa di PCR, se la concentrazione è superiore a questi parametri porta alla sintesi di prodotti aspecifici:
- [ Mg++ < 1 – 4 mM ] resa bassa.
- [ Mg++ > 4 ] porta alla sintesi di prodotti aspecifici.
- [Mg++ > 4 ] il Mg++ viene complessato con i dNTP inibendo la PCR.
Un tipico ciclo di PCR consta di tre fasi:
- Melting =separazione, denaturazione 94°C,
- Annealing = appaiamento dei primers 50°- 60°C,
- Elongation = estensione, allungamento 72°C.
Resa teorica della PCR : aumento esponenziale ad ogni ciclo:
2nxy dove
n = numero di cicli,
y = numero di copie iniziali di partenza.
Generalmente si compiono ≈ 30 cicli:
Esperienza di laboratorio 4, Tecniche di separazione:
Lo studio delle caratteristiche strutturali e delle proprietà di una sostanza richiedono tecniche di separazione e purificazione:
- Filtrazione,
- Distillazione,
- Centrifugazione,
- Precipitazione,
- Cromatografia.
Da cosa può essere rappresentato il materiale biologico?
Il materiale biologico può essere rappresentato da:
- Animali da esperimento,
- Un organo,
- Fettine di tessuto,
- Cellule isolate,
- Frazioni subcellulari (organelli),
- Macromolecole (proteine, acidi nucleici).
La centrifugazione
Isolamento della frazione subcellulare, Bisogna rompere la cellula usando un tampone che mantiene l’integrità dei componenti subcellulari. Il mezzo deve avere la stessa pressione osmotica del citoplasma in cui normalmente si trovano gli organelli.
Disintegrazione cellulare:
- Shock osmotico (globuli rossi in H2O),
- Congelamento/scongelamento,
- Digestione chimica o enzimatica della parete cellulare,
- Digestione meccanica,
La digestione meccanica consiste nell’omogeneizzazione e utilizza il:
- Waring-Blendor (frullatore), viene utilizzato per omogeneizzare grossi quantitativi di materiale di partenza.
- Potter- Elvehjeim, cilindro in cui si fa passare un pestello e si omogeneizza ancora più finemente l’omogeneizzato ottenuto con il Waring-Blendor.
Abbiamo inoltre sistemi meccanici basati sulla cavitazione (ossia producono cavità all’interno della membrana) basati sull’utilizzo di:
- Sonicazioni (ultrasuoni, 10000, 15000 Hertz),
- French press (pressione),
Materiale di partenza:
- Sospensione cellulare,
- Organo.
E’ necessario rompere la parete cellulare. Il risultato dopo digestione meccanica ottenuto con Waring-Blendor è che noi abbiamo ottenuto l’omogeneato cellulare (si è rotta la membrana cellulare e abbiamo gli organelli).
Come è possibile separare ciò che ci interessa?come riusciamo ad ottenere ciò? Mediante la centrifugazione.
La centrifugazione ci permette di separare il materiale che ci interessa. Al termine della centrifugazione otterremo un pellet che contiene ciò che ci interessa e un supernatante che elimineremo.
Centrifugazione differenziale con l’applicazione di campi centrifughi diversi. Prima della centrifugazione bisogna omogeneizzare. Lo si fa in ghiaccio per evitare l’attivazione di enzimi e si usa un tampone il cui pH sia il più vicino possibile al citoplasma Isolamento di mitocondri da fegato di ratto:
Raggio del rotore della centrifuga 11.5.
Rpm 4500.
Rcf 2600 (n.di giri).
Nel 1° pellet (dopo la prima centrifugazione) avremo materiale più pesante, che ha densità maggiore , ossia nuclei, materiale frammentato, cellule intere, acidi nucleici. A questo stadio non troveremo i mitocondri. Per pellettare i mitocondri dobbiamo separare il surnatante e centrifugarlo ad una velocità di rpm maggiore. In questa 2° fase si può ritrovare la frazione di mitocondri, ma non è pura (frazione cruda mitocondriale), perché vi si possono legare altri organelli che vengono tirati giù (purificazione blanda).
Per ottenere una purificazione spinta, si effettua una purificazione su gradiente. Allora la frazione mitocondriale sarà pura e si distribuirà secondo gradiente.
Dopo la prima fase in cui si è separato il surnatante dal pellet si ricentrifuga il surnatante a 12000 rpm. Quindi questa volta si usa il pellet che si forma e che contiene i mitocondri. Si aggiunge 500 μl di tampone e si passa tutto nel Potter e si ri-separa mediante centrifugazione. Si separa il surnatante e si utilizza il pellet che conterrà i mitocondri.

