Che cos’è una reazione a cetena della polimerasi
Il suo nome originale è Polymerase Chain Reaction, abbreviata in PCR.
Consiste in una metodica di Biologia Molecolare che permette l’amplificazione di una regione specifica di DNA mediante cicli ripetuti di replicazione a temperature differenti. La PCR è stata messa a punto negli anni ’80 da Mullis e collaboratori ed è stata applicata per la prima volta in medicina umana per la diagnosi prenatale di anemia falciforme mediante l’amplificazione del gene codificante per la ß-globina.
In cosa viene utilizzata la reazione a cetena della polimerasi
La PCR è estremamente specifica e sensibile: permette, infatti, l’amplificazione e l’identificazione di tratti genomici propri di un determinato agente patogeno (alta specificità) anche a partire da campioni biologici contenenti un numero esiguo di molecole bersaglio (alta sensibilità).
Il substrato per l’amplificazione (template) è rappresentato da un frammento di DNA a doppia catena (dsDNA).
Le metodiche attuali consentono di amplificare regioni di DNA lunghe fino a 10 Kba partire da una concentrazione iniziale di 1 nanogrammo di DNA. La PCR consente di amplificare sequenze di DNA provenienti sia da organismi unicellulari che da cellule aploidi o diploidi di organismi pluricellulari. È possibile amplificare il DNA anche se non è chimicamente puro, fermo restando l’assenza di inibitori della reazione. Risultati attendibili possono ottenersi impiegando la PCR anche su campioni contenenti DNA degradato come tessuti fissati in formalina o conservati in paraffina. I migliori risultati ovviamente si ottengono con campioni contenenti DNA integro, la cui presenza fornisce un template di qualità idonea per una PCR attendibile.
Su cosa si basa la reazione a cetena della polimerasi
La PCR si basa sul susseguirsi 3 di reazioni che avvengono a diverse temperature nella stessa provetta, con reagenti termostabili. Il volume totale della reazione nella provetta è – di 100 µ 25l: in aggiunta al campione (template) la PCR necessita di una DNA polimerasi, di 2 primers specifici, di oligonucleotidi (dNTPs) , di un sistema buffer – (TrisHCl e MgCl2 ), e di acqua bidistillata per portare a volume.
La DNA polimerasi che si utilizza correntemente è la Dna Polimerasi Taq , enzima termo-resistente isolato dal batterio Thermophilus aquaticus.
I primers forward e reverse, generalmente di lunghezza compresa tra 18 e 25 , bp dovrebbero a vere un contenuto di G+C non inferiore al -5060% e non devono essere formati da basi complementari per evitare che si appaino (dimerizzino) tra loro. Essi vengono sintetizzati da ditte specializzate in base alla sequenza di DNA alla quale si devono appaiare: il forward ha una sequenza identica alla sequenza 5’ -3’ del DNA template, mentre il reverse presenta una sequenza complementare (cioè è costituito da una sequenza di basi nucleotidiche complementari a quelle della sequenza target e con una direzione opp osta , ad es . la seq. del primer reverse CGGTCAAGTC si appaia alla seq. del DNA template GACTTGACCG ).
I deossiribonucleotidi trifosfati (dNTPs) forniscono le basi azotate per la sintesi del DNA.
Il buffer di reazione è composto da Tris – HCl e da MgCl2: i Tris – HCl tampona il H p della soluzione, mentre gli ioni Mg2+ sono cofattori fondamentali per l’attività della polimerasi.
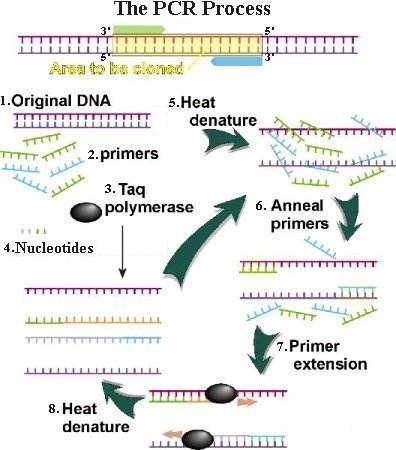
Le tre reazioni del ciclo di PCR
Per ogni ciclo si susseguono 3 reazioni:
- Denaturazione del template : avviene a -9296°C per 1’, temperatura in grado di rompere i legami idrogeno che uniscono le due eliche del DNA. Se il filamento di DNA è particolarmente ricco di G+C può essere necessaria l’aggiunta di glicerolo ed un incremento del tempo di denaturazione.
- Ibridazione o annealing : avviene solitamente a 50 -60 °C per 20”-1′. Ciascun primer si appaia all’estremità 3’ del filamento del DNA template, definendo così il tratto di DNA da amplificare.
- Estensione: avviene a 72°C per un tempo variabile in base alla lunghezza del frammento da amplificare: generalmente va da un minimo di 30’’ fino a -2’ . 1 La DNA polimerasi sintetizza un nuovo filamento di DNA in direzione 5’-3’ a partire dall’estremità libera del primer appaiato alla sequenza bersaglio : il DNA di nuova sintesi risulta essere complementare a lla sequenza del filamento denaturato.
Le 3 reazioni si susseguono in un numero variabile di cicli (generalmente – 3035) all’interno della stessa provetta. I cicli di amplificazione sono automatizzati ed avvengono in uno strumento detto termociclizzatore:
Poiché il DNA sintetizzato in ogni ciclo funge da stampo per la sintesi del ciclo successivo, la successione dei cicli comporta un aumento esponenziale del numero di copie di DNA: ad esempio, 20 cicli comportano un’amplificazione di circa un milione di volte.
Il DNA amplificato viene sottoposto a “corsa elettroforetica ” (60 -100 V) in gel di agarosio (1 – 2% di agarosio in tampone TBE): il DNA carico negativamente per la presenza dei gruppi fosfato migra dal polo negativo al polo positivo. Successivamente il gel viene immerso e colorato in una soluzione di etidio bromuro che si interpone fra le eliche del DNA e lo rende fluorescente ai raggi di un UV transilluminatore.
La prima “lane” o “pozzetto” a sinistra contiene il marker (cioè un DNA costituito da frammenti di peso molecolare noto), che consente di determinare la lunghezza (espressa in base pairs – bp) del frammento di DNA che si ottiene dall’amplificazione. Una maggiore intensità della banda di DNA è indice di una maggiore quantità dello stesso (espressa in nanogrammi – ng).