Pirosequenziamento
La rilevazione del nucleotide che viene incorporato si manifesta attraverso il rilascio di pirofosfato.
Fatto dall’università di Stoccolma nel 1996.
Estremamente rapido, elevatissima accuratezza e riproducibilità (99%), permette il real time sequencing con risultati che dalla processazione del campione al risultato finale ci vogliono solo 2 ore.
Tecnica partita con lo studio degli sNIP delle mutazioni in geni coinvolti con patologie tumorali o comunque in genetica umana.
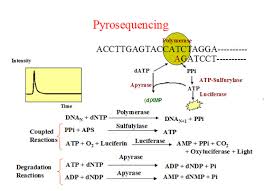
La cascata enzimatica nel pirosequenziamento
Abbiamo un frammento del quale vogliamo stabilire il contenuto di basi e questo contenuto nucleotidico è stabilito grazie alla presenza di una cascata enzimatica, ben 4 enzimi:
- Dna polimerasi
- Atp sulfurilasi
- la luciferasi che sarà quella che ci consente di visualizzare l’avvenuta reazione
- apirasi
Il frammento viene ibridizzato con un primer che serve da starter per la polimerasi.
La polimerasi incorporare un nucleotide, quando si è legato la sulfurilasi libera un pirofosfato per produrre ATP, l’ATP è il substrato della luciferasi che serve per produrre luce e manifestare l’avvenuta incorporazione.
Incorporato un nucleotide per volta, la pirasi degrada quei nucleotidi non incorporati, finchè non l’ha fatto non si aggiunge un ulteriore nucleotide.
Sembra un processo molto lungo in realtà avviene molto rapidamente.
Il pirogramma
L’output di questa reazione è il pirogramma ed è in relazione alla quantità di luce che viene prodotta dalla luciferasi e questo pirogramma altro non è che una serie di picchi e ciascun picco corrisponde ad una base che consentono di capire la sequenza del frammento preso in esame.
Ci vuole invece di 4-5 giorni delle normali procedure ci vuole solo 2 ore.
Ovviamente quel che serve è che il filamento stampo sia a singolo filamento.
Per fare ciò sono disponibili metodi commerciali molto rapidi che stabilizzano il single strand, in questo caso il sistema streptavidina/biotina in cui uno dei frammenti è biotinilato in cui una sferetta grazie alla presxenza della streptavidina lo cattura l’altro filamento viene degradato con NaOH e così nel pozzetto otteniamo un frammento a singolo filamento.
Le applicazioni, innanzitutto, il limite sta nella lunghezza dei frammenti da sequenziare al massimo 10-30 basi, si riesce a fare un real-time sequencing, nasce per le malattie genetiche nell’uomo mentre in microbiologia è stato applicato co nsuccesso per la tipizzazione del papilloma virus (agente eziologico del cancro alla cervice) o helicobacter pylori (coinvolto nell’insorgenza dell’ulcera peptica) per esempio per vedere le mutazioni che portano alla resistenza alla claritromicina il farmaco più comunemente usato al riguardo o ancora per studiare il virus dell’influenza.
Ci sono programmi che permettono di snellire l’analisi del sequenziamento, invece di vedere solo il cromatogramma ci permettono di effettuare anche ricerche in database, risolvere ambiguità, vedere le possibili sovrapposizioni tra i frammenti etc.
Per esempio Bioedit che non solo permette di leggere una sequenza ma anche di osservare il cromatogramma importante per lo studio degli eucariotici per quanto riguarda gli eterozigoti 8dove di solito mette N ma anche per problemi di backgound quidn ista all’operatore valutare caso per caso).
E se la sequenza è ancora sconosciuta usare BLAST che mi trovo l’omologia su tutto ciò che è nota ed in base all’omologia poterne capire la funzione o per valutare i domini conservati etc.

