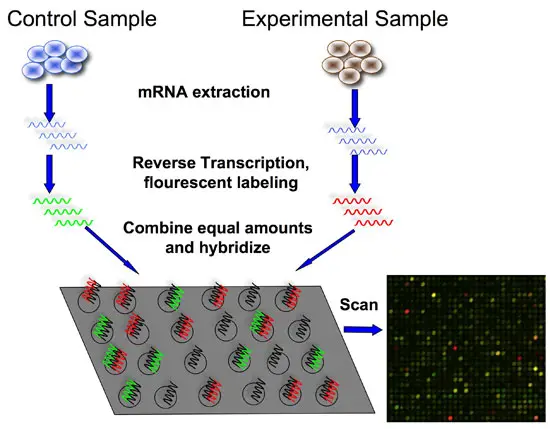
• Le sequenze nucleotidiche sono spottate.
• La loro lunghezza è di 25 basi.
• Non si usa una sola sonda per spot;
• Le sequenze nucleotidiche sono spottate.
• La loro lunghezza è di 25 basi.
• Non si usa una sola sonda per spot;
Il principio comunque è il solito di prima solo che userò c-DNA dell'RNA, perchè l'RNA è instabile e tende a ripiegarsi su sè stesso.
Quello che vedo dal DNA microarray devo semrpe confrontarlo con un'analisi di proteomica (elettroforesi) perchè non è detto che se un RNA è espresso 10 volte di più rispetto agli altri anche la proteina che ne deriva è espressa nella stessa quantità perchè l' RNA può essere degradato ci sono sempre le modifiche post-trasduzionali.
Quindi il limite principale è che non c'è un diretto controllo sull'espressione/traduzione della proteina.
Tecnica per studiare l'interazione tra patogeno-parassita in particolar modo durante l'infezione da parte di candida albicans, patogeno opportunista, nei monociti umani.
Cosa succede ai monociti quando vengono infettati.
E' seguito nel tempo la fagocitosi, e si vede che man mano che passa il tempo sale la fagocitosi, fino ad arrivare un plateau, perchè più di tanto i monociti non riescono a fagocitare.
Accanto c'è la mappa dei geni (rossi) sovraespressi in risposta all'aggressione da parte di Candica Albicans.
Tra i geni sovraespressi: geni dell'infiammazione, geni per la comunicazione delle cellule.
Altri geni sono down-regolati: geni per i recettori o fattori di trascrizione o tool like receptro.
C'è una mole di dati che posso valutarla con vari criteri per esempio temporale come in questo caso.
Così posso valutare i geni coinvolti nell'interazione ospite parassita.
Whole-cell array
E' del 2008 come argomento.
Il concetto dell'array cellulare è lo stesso di quello visto nelle lezioni passate con lo stesso principio degli array dove venivano immobilizzati su substrato degli oligonucleotidi delle proteine, noi andiamo a immobilizzare intere cellule possibilmetne vitali.
Queste cellule saranno in grado di interagire e noi saremo in grado di valutare l'avvenuta interazione con la stessa analogia con cui valutavamo l'interazione dna-dna oppure proteina-proteina.
valutare la risposta nell'insieme della cellula ad un determinato stimolo ambientale, ovviamente se poi questa cellula è anche eucariotica questa situazione ci consente di mimare quel che può succedere ad una cellula eucariotica superiore e all'estremo quel che accade nell'uomo (biochip).
le potenziali applicazioni sono quelle di creare dei veri sensori biologici, perchè ho una cellula che posso stimolare diversamente anche con stimoli tossici e valutarne la risposta applicazione farmaceutica magari un array di cellule batteriche per uno screening di molecole ad attività antibiotica, ma anche in ambito ambientale come valutare il livello di tossicità di composti presenti nell'ambiente e che possono essere messi in evidenzantramite varie risposti come cambiamenti morfologici.
La durata della tecnica può essere rapida ma quanto mi dura una cellula viva immobilizzata su substrato?
Questo è un suo limite, più di due settimane al momento è impossibile mantenere tali cellule vitali.
Le tecniche a disposizioni per valutare coem una cellula risponde:
• l'espressione dei geni in presenza di un composto
• evidenziare una determinata attività metabolica da parte della cellula
Non è molto economico perchè prevede l'impiego di apparecchiature motl osofisticate per spottarle, comuqneu una volta che i grossi costi sono ammortizzati con una produzione su vasta scala i costi sono bassi perchè non bisogna costruire sonde, non si ha bisogno di grossi reagenti, non c'è bisogno di ibridazione etc.
E' possibile anche posizionare non solo una piccola cellula ma motle cellule realizzare un array con un elevato un numero di cellule.
Gli array procariotici hanno altri vantaggi, meno si rapportano all'uomo come gli eucariotici, ma più semplici da maneggiare e modificare geneticamente.
Si vede l'array costruito dove sono depositate le cellule, l'array è sottoposto ad uno stimolo esterno, che può essere uno stimolo di stress, un agente tossico o un composto del quale vogliamo valutare la funzione.
E la rispostsa delle cellule che devono essere vitali, viene valutata in base alla:
• capacità di crescere
• se resistono allo stress e se vanno incontro a morte
• cambiamento morfologico che può essere molto importanti addirittura posson oessere presi come indice di vitaliatà della cellula dal punto di vista della trasduzione del segnale valutiamo:
• dimensione della cellula
• misurarle
• contarle
Se abbiamo costruito il nostro sistema cellulare, perchè costruito?
Perchè se abbiamo in qualche modo manipolato le nostre cellule batteriche che abbiamo ad esempio su un biochip batterico costruito su un vetrino possiamo anche averle rese in grado emettere fluorescenza e valutarne la risposta in questi termini oppure in un diverso metodo colorimetrico.
si possono analizzare i dati ottenuti e da qui definirne un pattern di risposta da parte della cellula allo stimolo al quale abbiamo sottoposto i nostri array.
Substrati: silicone, polimeri, fibre ottiche e nutrienti per crescere.
Le matrici dove sono posizionate o inglobate sono fatte di agar, alginato, polisaccaridi estratti da alghe che consentano loro di mantenersi.
Tra gli array delle cellule eucariotiche il vantaggio più importante è che da vicino simulano la risposta cellulare umana, poi di contro c'è più difficoltà ad effetturare manipolazioni perchè le cellule sono diplidi difficile creare mutanti perchè per arrivare ad essere knock-out per un gene ci vogliono almeno almeno a due cicli di trasformazioneil primo per ottenere un eterozigote il secondo per effettuare una delezione anche della seconda copia del gene.
Il meccanismo per posizionarle il cocnentto della stampante, cioè aghi che posizionano sulla superficie del vetrino un cellula o nei casi delle lezioni precedenti una proteina o un acido nucleico.
Altrimenti un metodo nuovo di posizionamento come il microcontact printing che prevede l'impiego di matrici d'alginato.
Gli array procariotici invece sono molto più semplici da creare, le cellule crescono apidamente, il primo array è stato l'E. Coli perchè cresce rapidamente, facile da manipolare, è resistente, è meno sensibile agli sbalzi ambientali.
Di tutto questo l'aspetto più importante per i procariotici è la facilità con cui può essere geneticamente modificato per consentirci di rilevare un determinato sengale.
I modelli più usati, nei procariotici, per valutare la risposta ad uno stimolo ambientale:
• uno light off: si attenuerà una risposta che posso quantificare come per esempio la luminescenza, alcuni batteri resi bioluminescenti, se c'è uno stimolo tossico ad esempio si riduce lo stimolo bioluminescente, molto meno usato;
• uno light on: aumenta la risposta, dipende da come abbiamo ingengnerizzato il microrganismo, il più semplice è quello di accoppiare la risposta di uno stimolo ad un promotore e ad un sistema rivelatore; per esempio per valutare un composto tossico posso pensare di fondere ai promotori che regolano la risposta allo stress un gene come la GFP o beta-galattosidasi o la fosfatasi alcalina valutando l'attività enzimatica o la resistenza ad u natibiotico che mi permette di visualizzare la risposta e il fulcro del disegno sperimentale è quello di scegliere quale promotore da fondere che sarà vincolata dal tipo di composto al quale voglio sottoporre il mio array per esempio se voglio valutare un composto che genera una risposta ossidativa andrò a scegliere il promotore che regola i geni che regolano la risposta ossidativa o geni per lo stress allo shock al calore; più usato;
Questi due sono i sistemi più utilizzati per l'allestimento e il biochip dei microchip procariotici.
Ovviamente non modifico solo un ceppo bensì un repertorio di microrganismi modificati in modo da valutare l'effetto di un composto in diversi background genetici (uno per lo stress ossidativo, uno per le heat shock protein, ripsosta a metalli pesanti etc.)
Un esempio del 2002.
Un array cellulare usando mutanti di E. Coli per valutare composti come i mono e disaccaridi.
Sempre nel 2002 usando un biosensore a cellule batteriche su unsubstrato a fibre ottiche.
Questo tipo di sistema nella pratica è un vetrino dove sono immobilizzate le cellule a livello di micropozzetti, le dimesnioni possono essere estese o ridotte a seconda del putno di vista fino ad arrivare a nano array, in ciascun pozzetto contiene un ceppo geneticamente modificato di una specie batterica ingengerizzato in modo tale da ripsondere ad u ndeterminato stimolo.
Un impiego è il ceppo di E.coli geneticamente modficiato prendendo come reporter il gene lac-z fuso al promotore di ZNTA che risponde alla presenza di metalli presenti, è un biosensore per rilevare la presenza di metalli pesanti.
Inoltre questo ceppo è ingegnerizzato in modo telae da presentare una proteina fluorescente che rende questa cellula in grado di emettere fluorescenza e rilevare la sua posizione all'interno dell'array.
Non è semplice interpretare i dati che provengono e a questo punto inizia la parte più difficile cioè la raccolta dei dati per valutare la "firma" dell'array la risposta specifica dell'array ad un determinato composto, nel caso di prima magari ci infilo un metallo pesante non ho solo la risposta di quel gene ma una serie di attività biologiche correlate.
Per arrivare ad avere l'array pronto magari ci vogliono anni per ottenerlo
Non esistono array disponibili nell'industria farmaceutica per molecole da analizzare e neanche nella diagnostica son tutti in fase sperimentale perchè il problema è mantenerli vitali.
Immaginatevi di avere da una massa di silicone ad un array con dei pozzetti ottenuto tramite varie procedure come quelle di manipolazione a livello mciromolecolare che eprmettono di ottenere i vari alloggiamenti.
Alcuni di questi polimeri possono avere difficoltà per esempio non essere sufficientemente trasparenti che magari non conviene per il metodo di rilevazione da utilizzare.
La problematica principale è quella del mantenimento.
Ecco alcuni sistemi di mantenimento per almeno 2 settimane.
La cellula deve essere vitale e comunque in grado di nutrirsi, se si pensa all'agar dopo 10 giorni lo spessore si assottiglia poi le colonie seccano e poi muoiono.
Difficile per una piastra immaginati in un microambiente.
Si usa per esempio il congelamento sottovuoto che potrebbe però danneggiare la cellula allora si pensa di incapsularla all'interno della sua matrice polimerica (agarosio, alginato, lattice, poliacrilammide etc.) che nel caso di array costruito con E. Coli le cellule rimangono vitali fino ad un massimo di 2 settimane.
Quindi gli ambiti applicativi ricapitolando sono:
• farmaceutico
• ambientale (per rilevarle in un ambiente o vedere se un molecola può essere tossica per l'ambiente)
Comunque sono tutti sistemi ancora in fase di ricerca.