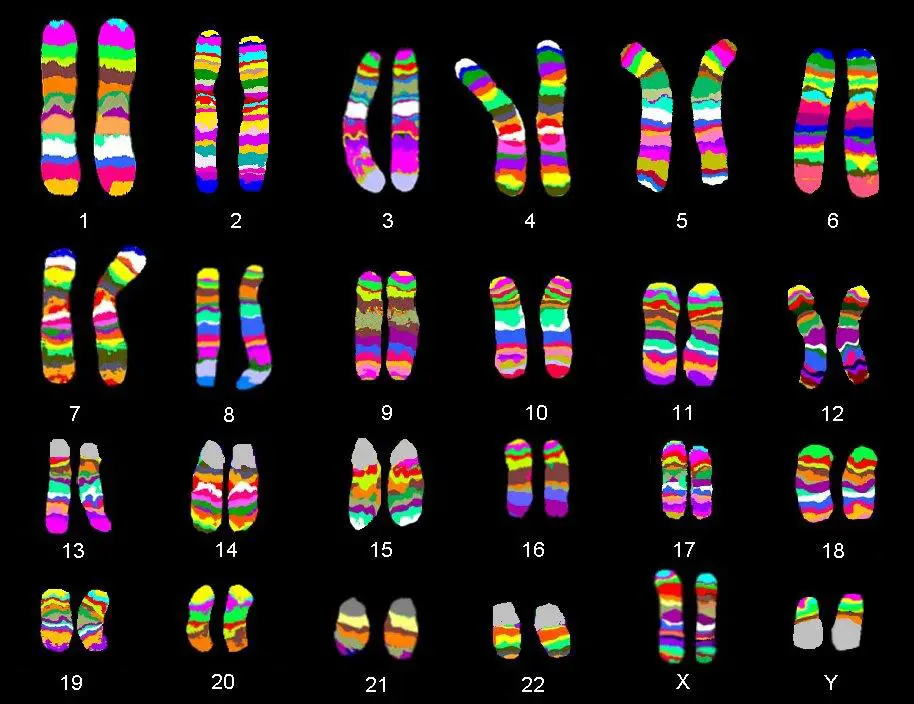
l'analisi del cariotipo si esplica tramite il seguente metodo. Molecole di dimensioni maggiori a 20Kb non vengono risolte con questa tecnica. L’assenza di risoluzione deriva dal fatto che per il DNA il rapporto carica/massa è costante.
Archivio Categoria: Biotecnologie Microbiche
È una disciplina che si propone di esplorare le potenzialità dell’utilizzo di microrganismi per diverse applicazioni biotecnologiche.
Tratta i diversi approcci metodologici mirati alla selezione ed al miglioramento genetico delle colture industriali, prende in considerazione esempi di produzione, caratterizzazione ed impiego dei principali prodotti delle fermentazioni industriali.
La sigla sta per "amplification fragment length polymorphism" quindi amplificazione di frammenti polimorfici ottenuti con la restrizione. E' una combinazione tra l'analisi di restrizione e la PCR. Quindi prima restrizione del DNA genomico del microrganismo da tipizzare con due tipi di enzimi di restrizion e poi un'aggiunta di molecole nucleotidiche chiamate adattatori che sono complementari […]
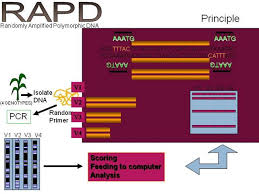
La sigla sta per "randomly amplified polymorphic DNA" • Amplificazione del DNA genomico “casuale” • Non è richiesta alcuna conoscenza “a priori” del genoma del microrganismo da tipizzare • Sequenza primer (un solo primer che dovrà legarsi ad entrambi i lati si lega sia in forward che in reverse) arbitraria molto corta, oligo 10 nucleotidi, […]
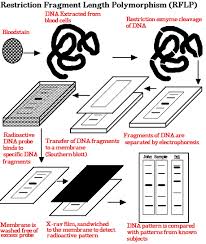
• dopo aver isolato il DNA genomica • digerisco tutto il genoma con enzimi di restrizione • migrazione elettroforetica Ha dei limiti perchè non riesco a distinguere bene i profili (bande tutte vicine vicine) con tutto il DNA genomico, quindi posso fare un RFLP riducendo il numero di bande trasferendo su un filtro di cellulosa […]
Parto dalle sequenze ITS che sono in banca dati. Si sceglie uno stretch di 30-35 paia di basi all'interno dell'ITS per ciascuna specie che sarà spottata sul vetrino. Facciamo in modo tale che la nostra sonda termini sempre una base prima dell'incorporazione della timina (si usa la tecnica APEX).
• Gene presente in tutti i batteri • Sequenze conservate • Regioni di eterogeneità • Database completo ed affidabile
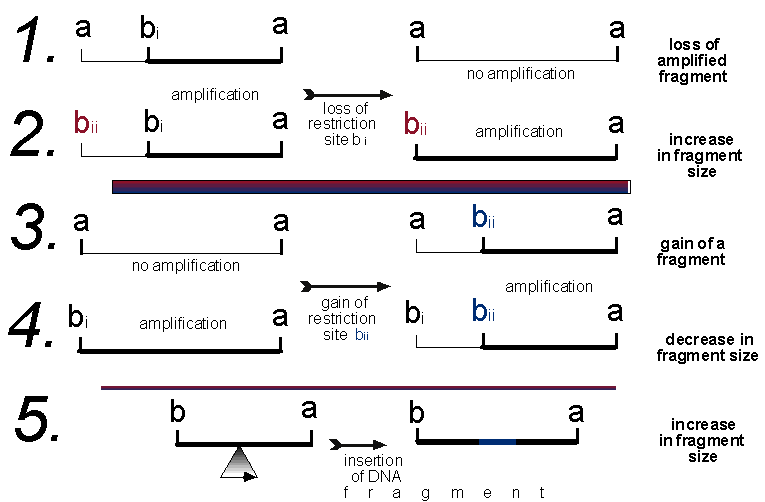
La particolarità è che si usa un primer disegnato in modo tale da avere un sito di restrizione quindi il primer si lega alla sua sequenza specifica, la DNA polimerasi allunga il primer, un enzima di restrizione taglia il primer in corrispondenza delle sequenze di riconoscimento del sito, la DNA polimerasi ripara il taglio e […]
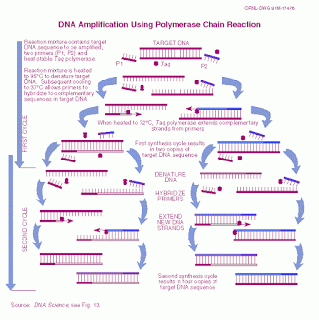
Di seguito descriviamo due metodiche su come amplificare il segnale in laboratorio, tramite target e tramite la sonda. AMPLIFICAZIONE DEL TARGET: Più intuitiva, si attua tramite una PCR quindi ha sensibilità elevata, un'elevata specificità, è rapida, può essere allestita mediante PCR convenzionale oppure modifiche della PCR classica tra cui la SDA.
• del target: più intuitiva, si attua tramite una PCR quindi ha sensibilità elevata, un'elevata specificità, è rapida, può essere allestita mediante PCR convenzionale oppure modifiche della PCR classica tra cui la SDA. Una volta effettuata la PCR si fà la verifica con il gel d'agarosio, la prsenza del bromuro d'etidio che ci permette di […]
Per una diagnostica di tipo molecolare dovremmo trovare il materiale genetico del microrganismo da identificare bisogna trovare sequenze target conservate ma variabili tra specie e specie sequenze specie-specifiche e si usano: